文章来源于互联网:新SOTA,AI增强医学蛋白质组数据分析,扩散模型驱动的从头肽测序
编辑 | 萝卜皮
随着人工智能工具的广泛应用,大多数技术和自然科学领域都在迅速发展。生物技术领域尤其如此,人工智能模型为药物研发、精准医疗、基因编辑、食品安全以及许多其他研究领域带来了重大突破。
其中一个分支领域是蛋白质组学,即大规模研究蛋白质的学科,该学科将大量蛋白质数据收集到数据库中,以便与样本进行比较。这些数据库使科学家能够辨别样本中存在哪些蛋白质(以及微生物)。它们使医生能够诊断疾病、监测治疗效果或识别患者样本中存在的病原体。
丹麦技术大学(Technical University of Denmark)生物工程系副教授 Timothy Patrick Jenkins 表示,虽然这些工具确实实用且有效,但它们的作用还是有限的:
「首先,没有哪个数据库可以包罗万象,所以你需要知道哪些数据库与你的特定需求相关。然后深度搜索非常耗时,需要消耗大量算力。最后,几乎不可能识别尚未被科学家探索的蛋白质。」
在这里,英国公司 InstaDeep、丹麦技术大学等组成的联合研究团队开发了 InstaNovo,这是一种将碎片离子峰转化为肽序列的转换模型。InstaNovo 的表现优于当前最先进的方法。
在此基础上,研究人员开发了 InstaNovo+,这是一种通过迭代细化预测序列来提高性能的扩散模型。使用它,研究人员提高了治疗测序覆盖率,发现了新的肽段,并在不同的数据集中检测了未报告的生物体,从而扩大了蛋白质组学搜索的范围和检测率。
该研究以「InstaNovo enables diffusion-powered de novo peptide sequencing in large-scale proteomics experiments」为题,于 2025 年 3 月 31 日发布在《Nature Machine Intelligence》。

基于串联质谱(MS/MS)的蛋白质组学主要依赖数据库搜索策略,该方法通过靶-诱饵库控制假发现率(FDR),但存在三大局限:
1、仅能识别预设数据库中的蛋白质序列,无法检测未知变异体、工程化序列或翻译错误;
2、修饰位点或非特异性酶切导致的搜索空间指数级增长会大幅增加计算成本与假阳性率;
3、固定数据库策略会遗漏剪接变体、单核苷酸多态性等潜在重要生物信息。
从头肽段测序作为数据库搜索的替代方案,通过前体离子碎裂图谱直接解析序列,特别适用于缺乏先验序列信息的蛋白质组学研究。深度学习推动了该领域的发展,但是计算成本呈线性增长,现有算法仍面临高错误率和性能不足的瓶颈,尚未完全突破大规模应用的限制。
InstaDeep、丹麦技术大学提出了一种使用多尺度正弦嵌入有效编码 MS 峰的变换模型 InstaNovo(IN),它在从头肽预测方面的表现超越了当下的 SOTA 方法,与现有工具相比,其准确率和召回率都有显著提高。
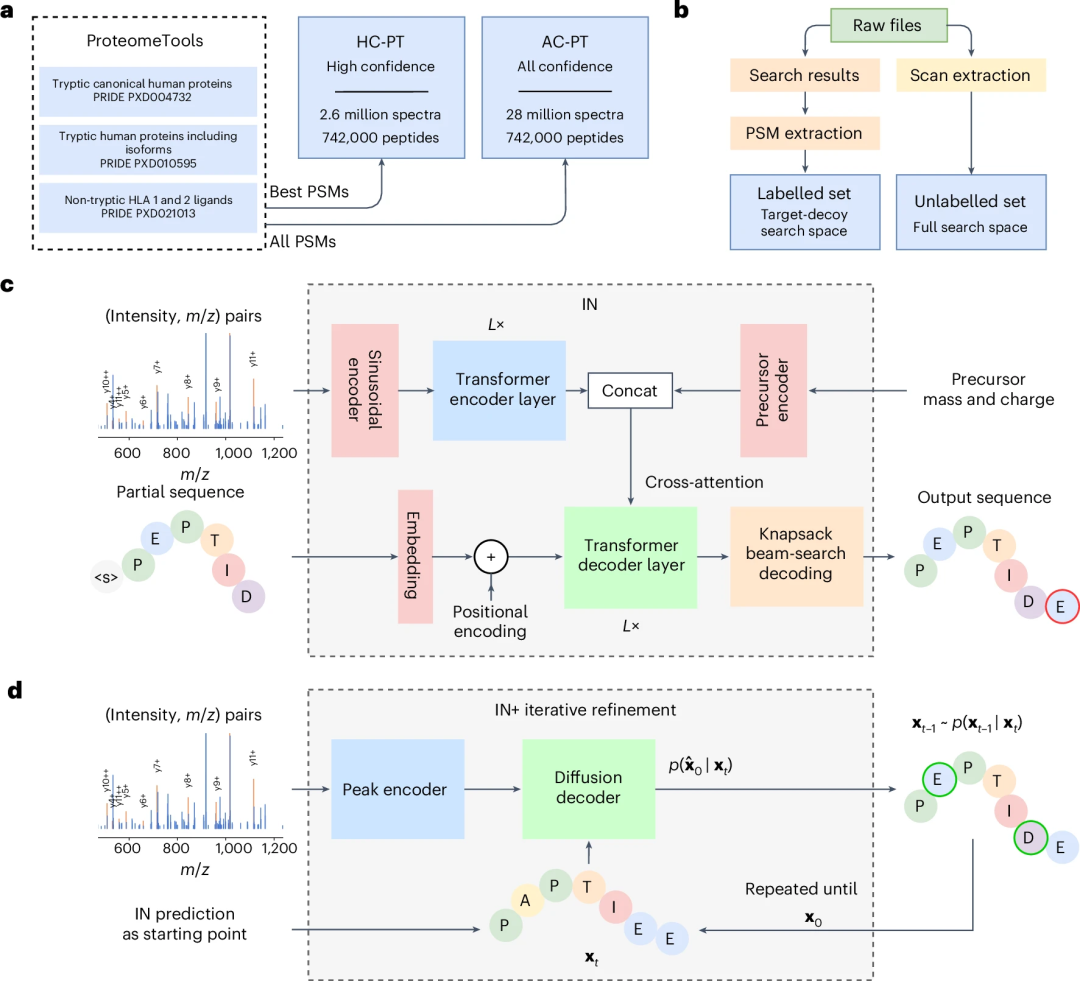
图示:InstaNovo 流程概述。(来源:论文)
研究人员在最大的可用蛋白质组学数据集 ProteomeTools 上训练了它。团队认为,Transformer 架构将很容易适应并适用于使用 MS 数据进行从头肽测序,因此他们设置了九个将质谱嵌入作为模型输入的 Transformer 解码器层来处理输入数据。研究人员应用 knapsack 集束搜索解码进行候选选择和肽评分。
基于扩散模型在谱图到序列预测中的优势,研究人员还开发了迭代精修模型 InstaNovo+,其通过 20 步迭代(结合时序嵌入的交叉注意力机制)逐步优化初始预测,能有效识别并修正 IN 模型的预测错误。
该模型编码器结构与 IN 相似,但通过结合质谱信息与肽序列更新的动态知识,实现了与人类专家解谱类似的「模糊预测-逐步精修」过程,验证集显示其性能显著提升。
研究人员将 IN 与当前最先进的模型 Casanovo 进行比较,同时还展示了在具体生物学问题中的应用。结果显示:
1、InstaNovo 为自下而上的蛋白质组学增加了价值和稳健性;
2、InstaNovo 可检测 HeLa 细胞中一半以上的人类蛋白质组,并扩大新型生物制剂的序列覆盖范围;
3、InstaNovo 在蛋白质组中发现新蛋白质和病原体;
4、InstaNovo 识别免疫肽组和降解组中的肽。
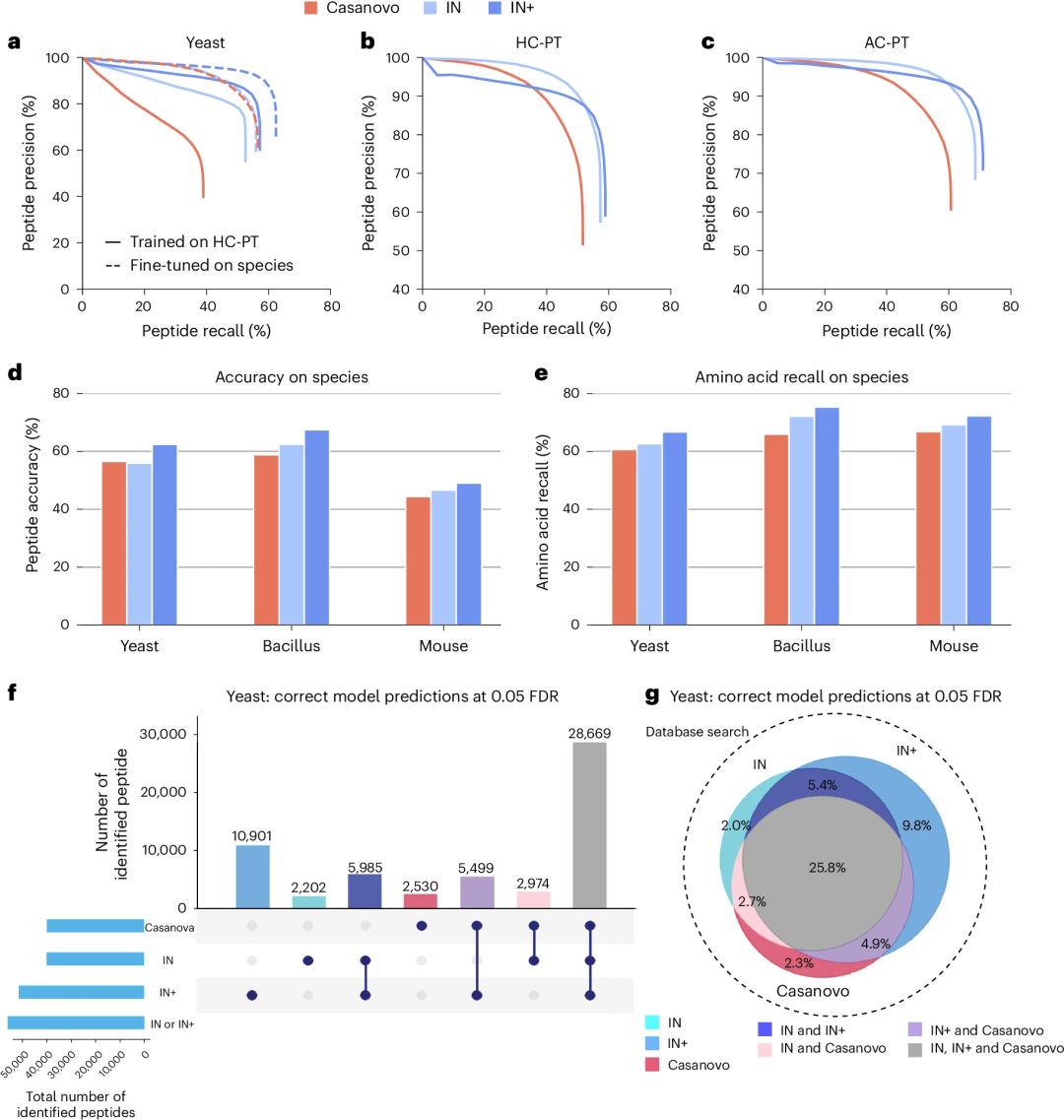
图示:Casanovo、InstaNovo 和 InstaNovo+ 的比较评估。(来源:论文)
「综合来看,我们的模型超越了最先进的水平,并且比目前可用的工具精确得多。此外,正如我们在论文中所展示的,我们的模型并不针对特定的研究领域。相反,这些工具可以推动涉及蛋白质组学的所有领域的重大进步。」InstaDeep 的研究工程师、论文的共同第一作者 Kevin Michael Eloff 说。
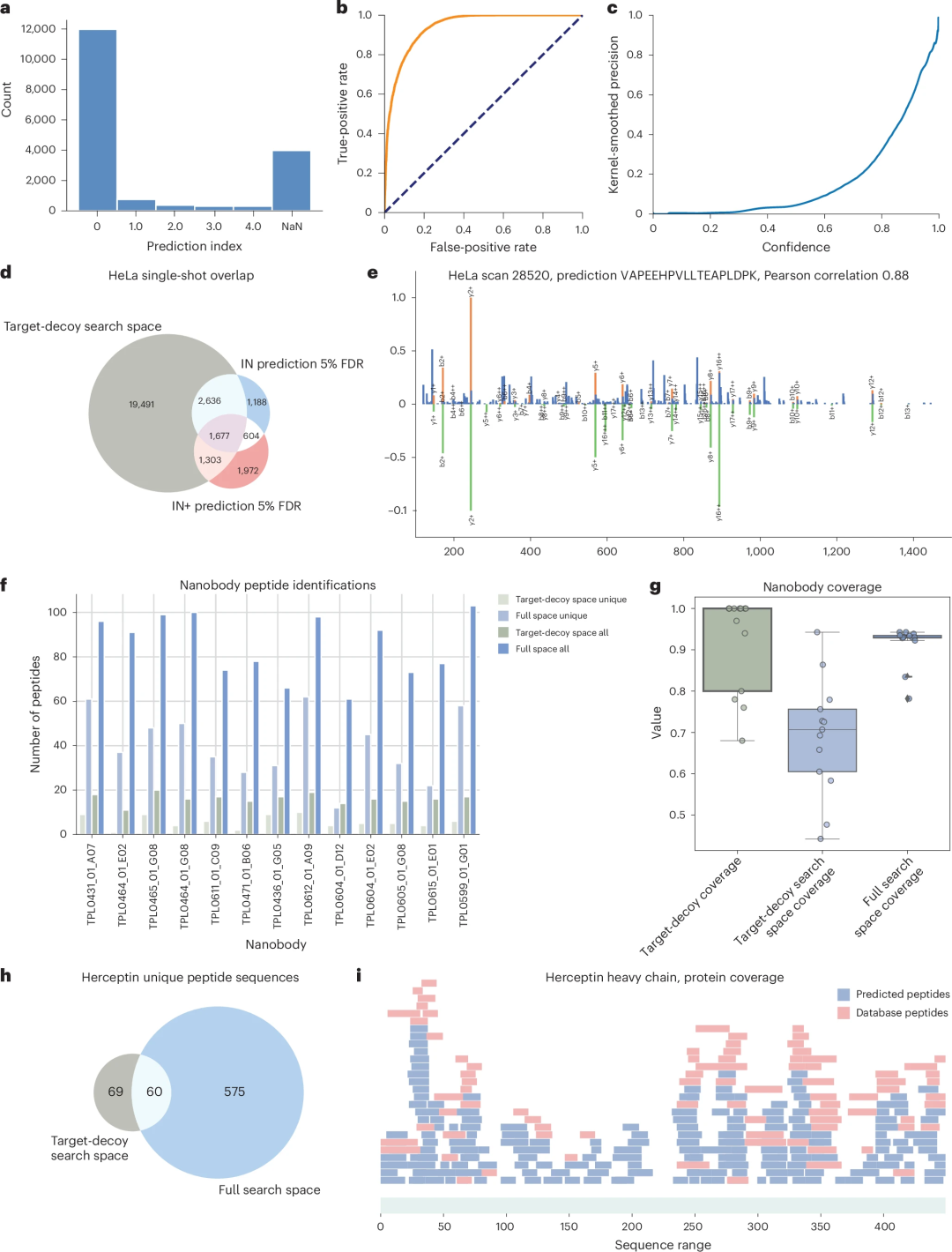
图示:InstaNovo 在已建立的 HeLa 蛋白质组和不同格式的序列治疗上实现了良好的准确性。(来源:论文)
具体来说,研究人员在八个不同的数据集中实现了新的生物学发现,包括识别 HeLa 细胞中数据库搜索未检测到的蛋白质、将免疫肽组学数据集扩展了 175% 以上的肽以及表征了新的蛋白水解裂解。
研究人员在论文里表示:「鉴于我们的结果和本研究中探索的数据集的多样性,我们预计该模型可以在生物体和生物样本中具有高精度和令人满意的性能。」
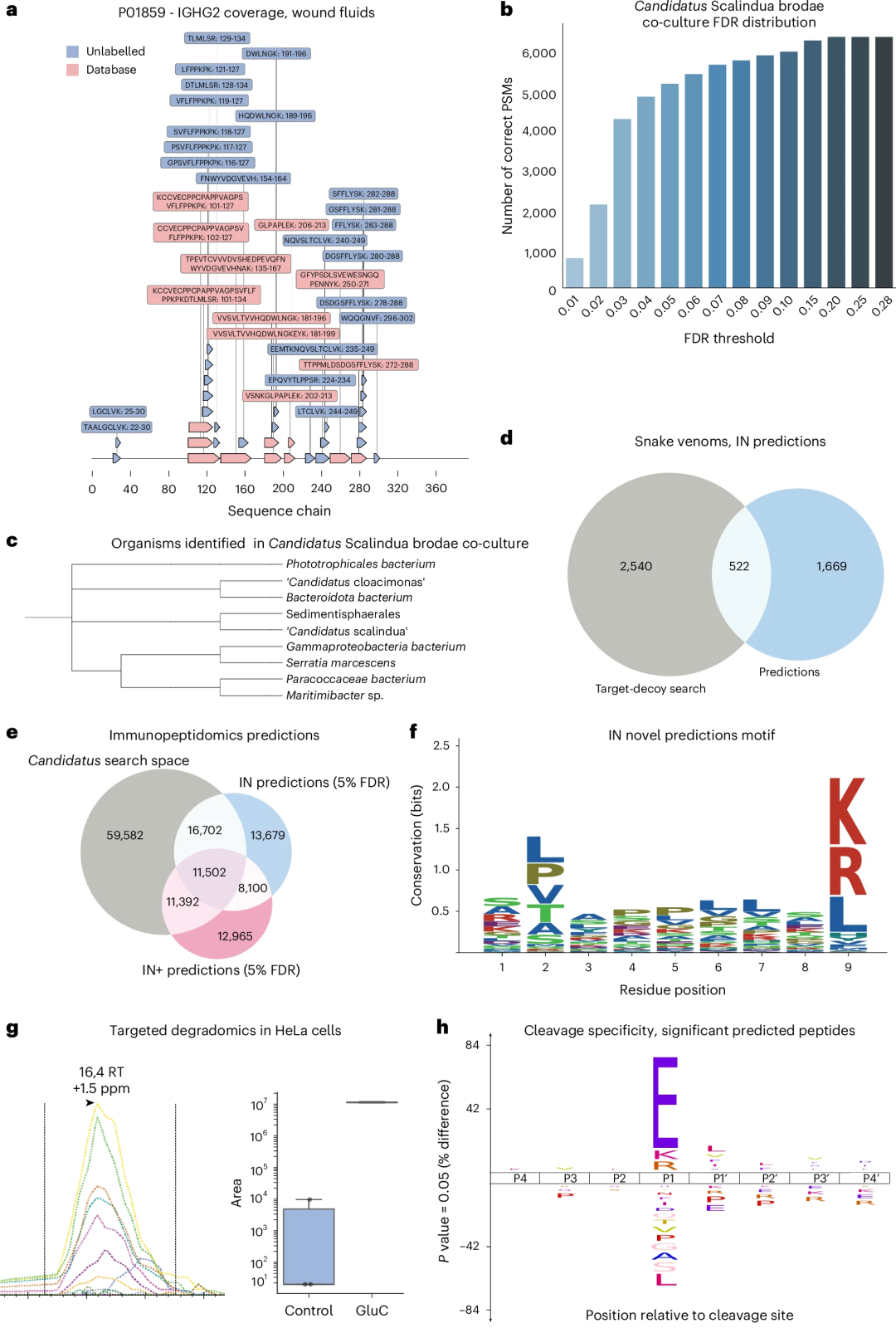
图示:InstaNovo 增加了蛋白质覆盖率,识别了新生物,并检测了半胰蛋白酶肽和非胰蛋白酶肽。(来源:论文)
该团队认为该模型未来将应用于更广泛的研究领域,例如蛋白质组学、肠道微生物组研究和旨在探索未报告蛋白质形式的研究。他们还希望 IN 能够在单细胞蛋白质组学这一新兴领域找到合适的应用。
团队表示:规模是决定从头肽测序模型性能的最重要因素,就像使用 Transformer 架构的其他领域一样。他们希望通过利用存储库中提供的大量 MS 数据集来进一步提高模型性能。
研究人员还期待同行可以广泛尝试他们的技术,并期待进一步探索微调、蛋白质推断和组装,以及在该基础模型之上构建用于混合或从头搜索的应用程序。
开源代码:
https://github.com/instadeepai/InstaNovo
https://doi.org/10.5281/zenodo.14712453
9 个物种的数据集:
https://huggingface.co/datasets/InstaDeepAI/ms_ninespecies_benchmark
高置信度ProteomeTools 数据集:
https://huggingface.co/datasets/InstaDeepAI/ms_proteometools
数据分析和可视化的自定义脚本:
https://doi.org/10.6084/m9.figshare.24173889.v1
论文链接:
https://www.nature.com/articles/s42256-025-01019-5
相关报道:
https://phys.org/news/2025-03-ai-protein-analysis-medical.html
文章来源于互联网:新SOTA,AI增强医学蛋白质组数据分析,扩散模型驱动的从头肽测序


